AG Staiger
Institut für Neuroanatomie
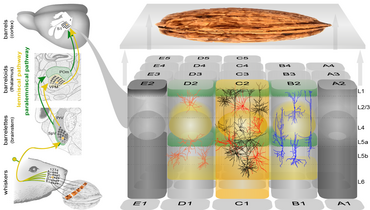
Kolumnen können als Grundeinheit für die kortikale Informationsverarbeitung angesehen werden. Wir interessieren uns für die Verarbeitung taktiler Informationen im primären somatosensorischen Kortex, wo Barrel-assoziierte Säulen bei Nagetieren besonders gut identifiziert werden können. Aufgrund der ausgeprägten Heterogenität von Neuronen in einer kortikalen Säule scheint der Ansatz, die funktionelle und strukturelle Konnektivität einzelner, molekular definierter Neuronen in vitro und in vivo zu untersuchen, besonders vielversprechend für die Identifizierung von Grundprinzipien der säulenförmigen Informationsverarbeitung.
Unter Verwendung des Barrel-Cortex der Maus als Modellsystem (siehe Abbildung) arbeiten wir an mehreren Fragen zur Konnektivität erregender und inhibitorischer Neuronen, um den Bauplan kortikaler Mikroschaltkreise zu entschlüsseln.
Unsere aktuelle Forschung nutzt transgene Mausmodelle, um Zugang zu genetisch markierten Neuronen zu erhalten, was uns ermöglicht, vorab identifizierte Subpopulationen GABAerger Neuronen gezielt zu untersuchen. Da frühere transkriptomische Studien auf mehr als 40 Arten GABAerger Neuronen schließen ließen, nutzen wir nun auch die intersektionale Mausgenetik, um einen immer spezifischeren Zugang zu diesen Neuronen zu erhalten. Grundsätzlich wollen wir die neuronale Identität und Funktion durch multimodale Charakterisierung von (i) Transkriptom, (ii) Morphologie, (iii) Elektrophysiologie und (iv) Konnektivität „entschlüsseln“. Die Methoden der Wahl sind transgene Mäuse, Patch-seq und gepaarte Aufnahmen in Kombination mit 3D-Rekonstruktionen von Biocytin-gefüllten Zellen.
Diese leistungsstarke Methodenpalette wurde kürzlich auch auf Primatenmaterial angewendet. Durch die Einbeziehung von Weißbüschelaffen und Makaken in unsere Forschungsrichtung sind wir in der Lage, Zelltypen zwischen Primaten- und Nicht-Primatenarten direkt zu vergleichen.
Für einen tieferen Einblick in unsere Forschung schauen Sie sich bitte unsere Veröffentlichungen an.